实验环境
本实验基于huggingface/transforms-PyTorch框架进行词语级别的语义相似度计算。实验环境使用Windows操作系统,涉及bert-base-multilingual-uncased和biobert-base-cased-v1.1两个预训练模型,具体实验配置如下表所示。

模型介绍
本次实验总共涉及两个模型,即Bert和BioBert。相关的模型介绍如下。
Bert模型[1]通过在所有层的上下文联合调节来预训练深层双向Transformer编码器,作为一种新型的语言模型,融合了基于特征和基于微调的任务策略,在诸多下游任务上取得了最优的效果,通过论文的消融研究发现Bert模型的双向性质对模型的改进起了很大的作用。Bert模型目前实现了两个版本,Base版本拥有12层、768个隐藏大小、110M的自注意力数量,Large版本拥有24层、1024个隐藏大小、340M的自注意力数量。
BioBERT模型[2]是用于生物医学文本挖掘的双向编码器表示Transformers,该模型使用和Bert相同的结构,在大型生物医学语料库上进行了训练, 作为第一个基于领域特定BERT的模型,它能够很好的处理生物医学领域的文本。
实验过程与结果
原理分析
计算词语级别相似度的实验过程可以大致分为两个阶段,首先基于预训练模型获取词向量,进而使用特征表示向量在向量空间上进行相似度计算。
在获取词语级别的特征表示向量阶段。两个实验均采用Bert模型结构,以词语作为最小单位进行输入,模型针对输入的词语根据词汇表和内置的WordPiece模型进行tokenize,部分未在模型中出现的生物医学领域词汇将会被分割成多个子词。通过预训练模型可以获得每个子单词不同层面的上下文表达语义向量,最终选择取最后一层输出的隐含层状态序列并对子词Embedding求平均得到词语级别的向量。经过计算后每个词语都将对应一个768维的特征向量用于后续计算,具体核心代码如下。
encoded_input=tokenizer(word,return_tensors='pt')
output=model(**encoded_input)
embeddings.append(torch.mean(output.last_hidden_state[0,:,:], dim=0))
在计算语义相似度阶段。使用较为常用的Cosin相似度作为最后的相似度评估标准。
sim(S,T)=(S^T T)/‖S‖‖T‖
对每个词语的特征向量两两间求相似度,具体核心代码如下。
def cosine_sim(x,y):
num=sum(map(float,x*y))
denom=np.linalg.norm(x)*np.linalg.norm(y)
return num/float(denom)
实验1
可以观察到词汇“rötheln”属于德语词汇,其他的词汇(“german measles”,“rubella”,“morbilli”,“rubeola”)均属于英语词汇,而用于计算相似度的BioBert预训练模型大部分基于英文语料库进行训练。因此如果在之后的实验中选择使用“rotheln”代替“rötheln”进行进一步的相似度验证,就需先判断这两个词汇是否属于同源词汇。
实验使用bert-base-multilingual-uncased预训练模型,该多语言模型选取Wikipedias 中 top104 的语言语料库进行训练,并且采用指数加权平滑对不同语种进行了采样,可以处理包括德语在内的104种语言文本。实验以词语“rötheln”和“rotheln”作为输入,最终得到两者的语义相似度为1.0000004768371582。
实验2
实验使用biobert-base-cased-v1.1预训练模型,该模型在原Bert基础上添加了大型生物医学语料库PubMed和PMC的训练。本实验基于transforms接口获取模型以及分词器,以词语(“german measles”,“rubella”,“rötheln”,“rotheln”, “morbilli”,“rubeola”)作为输入。通过BioBert获得对应词汇的特征向量后,使用余弦相似度两两计算词汇之间的相似度,最后选择matplotlib包对相似度结果矩阵进行可视化。最终的语义相似度结果如下图所示。
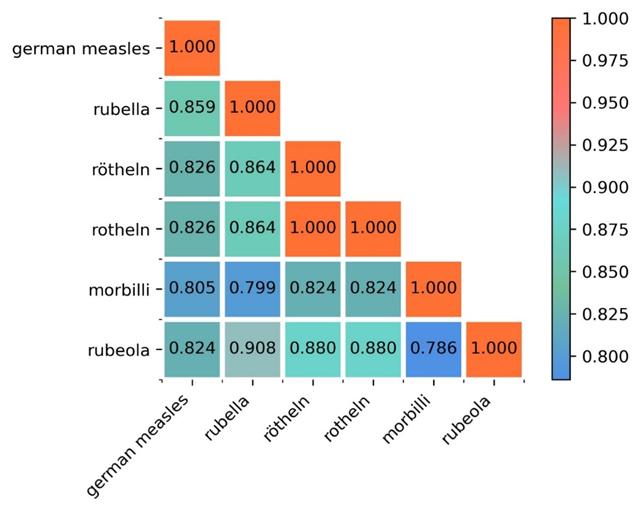
实验结果分析
通过实验发现“rötheln”和“rotheln”的语义相似度为1,属于同源词汇。分析“german measles”与其他几个词汇的相似度可以发现“rubella”与其语义相似度最高,达到了85.9%。剩下几个词汇与其语义相似度从高到低排序依次为“rötheln”,“rotheln”,“morbilli”,“rubeola”。
[1] Devlin J, Chang M W, Lee K, et al. Bert: Pre-training of deep bidirectional transformers for language understanding[J]. arXiv preprint arXiv:1810.04805, 2018.
[2] Lee J, Yoon W, Kim S, et al. BioBERT: a pre-trained biomedical language representation model for biomedical text mining[J]. Bioinformatics, 2020, 36(4): 1234-1240.